Вариабельность ДНК спутника pBuM189 среди южноамериканских популяций Drosophila buzzatii
Машинный перевод
Оригинальная статья написана на языке EN (ссылка для ознакомления) .
ДНК спутника pBuM189 была проанализирована в популяциях Drosophila buzzatii, которые охватывают большую часть ареала вида в Южной Америке. Этот satDNA состоит из богатых A+T мономеров длиной 189 п.н. и предыдущие исследования показали быстрый темп эволюционных изменений этого компонента генома D. buzzatii. Всего было изучено 63 единицы повторения pBuM189 из 14 популяций D. buzzatii (9 из Бразилии и 5 из Аргентины). Средняя нуклеотидная изменчивость среди 63 повторов составляет 4,2%. По крайней мере, один повтор (Juan/4) кажется частью другой подсемейства satDNA pBuM189. Нуклеотидное выравнивание всех 63 повторов не выявило специфических нуклеотидных замен или инделов, которые могли бы различать каждую популяцию или группы географически близких популяций. Такое отсутствие интерпопуляционной дифференциации satDNA согласуется с предыдущими данными по mtDNA, которые указывают на высокий генетический поток и очень небольшую дифференциацию популяций на протяжении большей части ареала D. buzzatii в Южной Америке. Генетический поток мог быть возможен во время ледниковых событий в плейстоцене, таких как то, что произошло между 13000 и 18000 лет назад, когда D. buzzatii вероятно имел более непрерывное распределение, чем то, что наблюдается сегодня.
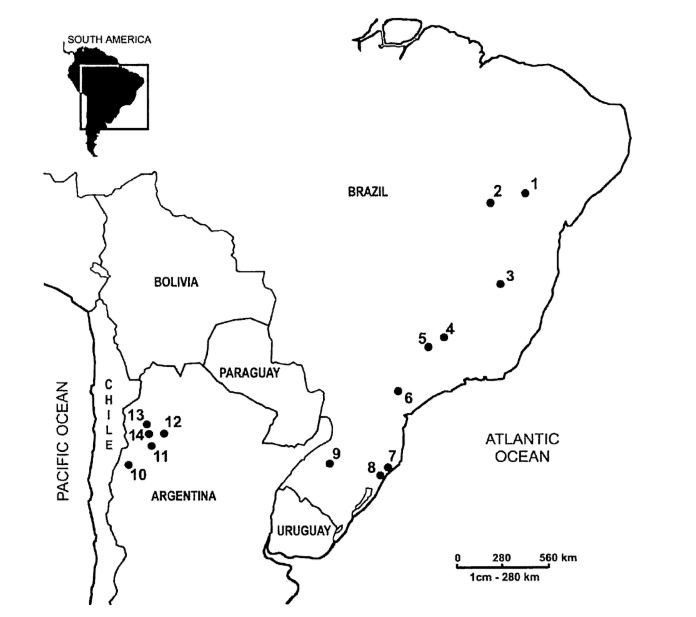
Кактусофильная муха Drosophila buzzatii (buzzatii кластер, repleta группа) имеет широкое распространение в Южной Америке, встречаясь на большинстве территорий Аргентины, Боливии, Парагвая и Бразилии. Этот вид использует разлагающиеся ткани нескольких кактусов опунции (Opuntia spp.) и колонновидных кактусов для питания и размножения (PEREIRA et al. 1983). За последние двести лет кактусы Opuntia были перенесены человеком в несколько регионов мира, включая страны Средиземноморья, Африки и Австралии. В результате D.buzzatii мигрировала вместе со своими хозяевами. Эта ситуация сделала D.buzzatii подходящим организмом для изучения вопросов, связанных с генетическими изменениями в процессе колонизации (FONTDEVILA et al. 1982; HALLIBURTON and BARKER 1993; ROSSI et al. 1996; FRYDENBERG et al. 2002).
Согласно данным о хромосомных инверсиях, аргентинский Чако является наиболее вероятным центром ее радиации (WASSERMAN 1962; CARSON and WASSERMAN 1965). D.buzzatii также очень распространена в Чако, ассоциируясь с рядом различных видов кактусов (Vilela et al. 1980). В общем, бразильские популяции распределены пятнами по небольшим участкам ксерофитной растительности, где они встречаются с очень низкой частотой (VILELA et al. 1983; TIDON-SKLORZ et al. 1994; TIDON-SKLORZ and SENE 1995). Единственным исключением является южная Бразилия, где были найдены относительно крупные популяции (VILELA et al. 1983; RUIZ et al. 2000).
Предполагается, что D. buzzatii колонизировала Бразилию через изменения в распределении аридной растительности во время ледниковых периодов, таких как те, что произошли в позднем плейстоцене (BARKER et al. 1985; FIGUEIREDO and SENE 1992).
BAIMAI et al. (1983) проанализировали метафазные хромосомы нескольких популяций D. buzzatii из Аргентины и Бразилии. Все исследованные популяции демонстрируют одинаковую метафазную пластинку. BARKER et al. (1985) не обнаружили значительных аллозимных различий среди южноамериканских популяций. Только небольшая локальная дифференциация, похоже, произошла в бразильских популяциях из Баии (северо-восток) и Траманда´1 (юг). В другом исследовании FIGUEIREDO и SENE (1992) сообщили, что из 16 хромосомных инверсий, обнаруженных в Аргентине, только две были представлены в бразильских популяциях, и ни одной не было найдено на северо-востоке Бразилии. ROSSI et al. (1996) и DE BRITO et al. (2002) обнаружили высокие уровни однородности mtDNA среди южноамериканских популяций D. buzzatii и несколько тестов показали высокий генетический поток на протяжении большей части географического распределения D. buzzatii . DE BRITO et al. (2002) также подсчитали, что D. buzzatii присутствует в Бразилии как минимум 100 тысяч лет, что предполагает предголоценовое расширение популяций D. buzzatii из Аргентины в сторону Бразилии.
Чтобы получить больше информации о генетической изменчивости D. buzzatii в Южной Америке, мы искали ядерную ДНК. Сателлитная ДНК (satDNA) — это класс высокорепетитивной некодирующей ДНК, которая организована в большие однородные массивы в геноме почти всех эукариотических организмов. Обычно быстрый темп эволюционных изменений последовательностей satDNA поддерживается рядом семейств satDNA, которые оказались специфичными для видов (BACH-MANN et al. 1989; KING and CUMMINGS 1997).
Предыдущие исследования показали, что последовательности satDNA могут быть полезны для различения консpecific популяций (ELDER and TURNER 1994; WU et al. 1999).
KUHN et al. (1999) описали сателлитную ДНК pBuM189 D. buzzatii. Она состоит из повторяющихся единиц, слегка богатых AT и длиной 189 п.н. Пять описанных последовательностей pBuM189 были получены из геномной ДНК только одной популяции D. buzzatii. Гибридизация и эксперименты ПЦР не смогли обнаружить последовательности pBuM189 в геноме близкородственных видов, таких как D. serido, D. borborema или D. koepferae. Поэтому имеющиеся данные указывают на быстрый темп эволюционных изменений этого компонента ядерной ДНК D. buzzatii.
В настоящем исследовании мы проанализировали 63 последовательности pBuM189 из штаммов, представляющих 14 популяций D. buzzatii (9 бразильских и 5 аргентинских). Популяции охватывают большую часть географического распределения этого вида в Южной Америке.
Материалы и методы
Образцы
Географическое расположение всех sampled популяций D. buzzatii показано на рис. 1. Используемые штаммы с их соответствующими местоположениями: D69R5 (1); J79H41 (2); D54F5 (3); H86G8 (4); D42F2 (5); H99X6 (6); H42F1 (7); J25A20 (8); J28E15 (9); ArgE3 (10); Cat (11), Hondo (12); Salta (13) и Ticucho (14).
Молекулярные техники
Последовательности pBuM189 были изначально выделены из генома D. buzzatii после дигестации ДНК с помощью фермента MspI (KUHN et al. 1999). В настоящей работе был использован фермент SspI (который также присутствует только один раз на единицу повтора pBuM189), чтобы облегчить процедуру клонирования. Фрагменты satDNA pBuM189 были идентифицированы по их появлению в виде ярких полос примерно 190 п.н. на фоне размытости геномной ДНК. ДНК из полосы ДНК 190 п.н. была элюирована из геля путем инкубации на ночь в 500mM NaAc; 1mM EDTA. Восстановленные фрагменты были лигированы в вектор плазмиды pUC18 и трансформированы в компетентные клетки Escherichia coli DH5-a. Рекомбинантные клоны были отобраны в виде белых колоний на ампициллиновых пластинах, содержащих X-gal (5-бромо-4-хлоро-3-индолил-β-D-галактозид) и IPTG (изопропил-β-D-тиогалактопиранозид). Реакция шаблонной ДНК для секвенирования была подготовлена согласно инструкции набора BigDye Terminator Cycle Sequencing Ready Reaction Kit (Perkin-Elmer). Автоматическое секвенирование ДНК было выполнено на секвенаторе ABI Prism™ 377 (Perkin-Elmer).
Для оценки нуклеотидной изменчивости, генетических расстояний и построения NJ дендрограммы использовалась программа MEGA 2.1 (KUMAR et al. 2001).
Результаты и обсуждение
Как уже ожидалось из ранее полученных данных последовательности pBuM189, дигестация геномной ДНК 13 штаммов D. buzzatii с SspI дала сильно окрашенную ДНК полосу примерно 190 п.н., соответствующую мономерам pBuM189, за которой следовали полосы с уменьшающейся интенсивностью сигналов 380 п.н. (димеры), 570 п.н. (тримеры) и так далее. Полоса ДНК 190 п.н. из каждой дигестации была независимо элюирована из геля и клонирована. Всего было секвенировано 58 клонов, 39 из 8 бразильских популяций (местоположения 1 – 4, 6 – 9) и 19 из 5 аргентинских популяций (местоположения 10 – 14). Нуклеотидное выравнивание этих 58 клонов с 5 повторами pBuM189 (sert/1 – 5 в настоящей работе) из местоположения (5), описанного ранее KUHN et al. (1999), подтвердило, что все они представляют собой мономеры satDNA pBuM189 (Рис. 2).

63 повтора pBuM189 слегка обогащены A+T (71 %) и средняя изменчивость последовательности составляет 4,2 %. Основным источником вариации среди 63 повторов pBuM189 являются однонуклеотидные замены, из которых 69 % специфичны для повтора, а оставшиеся общие для двух или более повторов. Было найдено только 11 инделов.
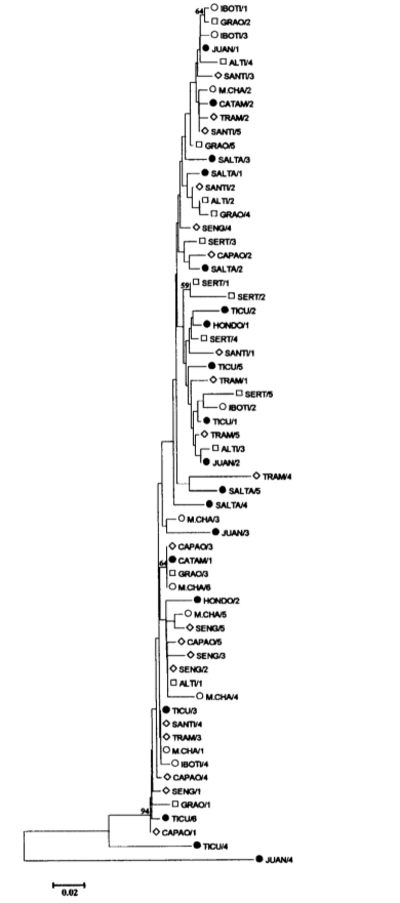
Один повтор pBuM189 кажется исключительным. Клон Juan/4 из местности (10) продемонстрировал неожиданно высокие уровни дивергенции последовательности во всех парных сравнениях (в среднем 22,1 % и всегда превышая 19 %). Более того, в то время как типичные последовательности pBuM189 имеют в среднем 1,7 % специфичных для повтора однонуклеотидных замен, в повторе Juan/4 было найдено 30 (например, T на позициях 11 и 42). Такой высокий уровень нуклеотидной дивергенции предполагает, что повтор Juan/4 может принадлежать другой подсемье pBuM189. С теоретической точки зрения, происхождение подсемей satDNA зависит от таких переменных, как число копий, геномное распределение семейства satDNA и относительные скорости нерециркулярных обменов внутри и между не гомологичными хромосомами (DOVER 1986).
Если повтор Juan/4 исключить из выборки, средняя нуклеотидная изменчивость оставшихся 62 последовательностей pBuM189 уменьшается до 3,7 %. Это значение почти идентично 3,8 %, рассчитанному KUHN и др. (1999) на основе пяти последовательностей pBuM189, изолированных только из одной популяции D. buzzatii (местоположение 5).
Среди других повторов pBuM189, Ticu/4 из местоположения (14) наиболее похож на Juan/4, так как содержит два специфических нуклеотида (G на позиции 70 и A на позиции 88) и один индел (позиция 49), которые в противном случае являются эксклюзивными для повтора Juan/4, а также несколько других замен, общих для Juan/4 и других повторов (например, T на позиции 36 или A на позиции 133). Сам повтор Ticu/4 является наиболее дивергентным повтором pBuM189 (в среднем 10,2 %) после Juan/4, представляя 6 специфических замен нуклеотидов (позиции 1, 69, 79, 97, 98, 167 и 188). Таким образом, Ticu/4 следует рассматривать как промежуточный эволюционный этап между «типичными» повторами pBuM189 и сильно дивергентным повтором Juan/4. Интересно, что оба редких повтора Juan/4 и Ticu/4 происходят из Аргентины, где, вероятно, возник вид (WASSERMAN 1962; CARSON и WASSERMAN 1965).
Согласно теоретическим и эмпирическим данным, дифференциация последовательностей satDNA должна происходить в соответствии с концепцией согласованной эволюции (DOVER 1986; ELDER и TURNER 1994). Другими словами, массивы satDNA из разных популяций или видов могут быть гомогенизированы для различных мутаций под воздействием молекулярных механизмов, таких как неравномерный кроссинговер и конверсия генов, связанные с генетической изоляцией и генетическим дрейфом.
Нуклеотидное выравнивание 63 повторов pBuM189 из 14 популяций D. buzzatii (в среднем 4,5 % повторов на популяцию) не выявило специфической нуклеотидной замены или индела, которые могли бы различать конкретную популяцию или группы географически близких популяций (Рис. 2). В некоторых случаях более двух клонов имели идентичную нуклеотидную последовательность, как в случае клонов Capao/3, Grao/3, M.Cha/6 и Catam/1 или M.Cha/1 и Santi/4, но они происходят из довольно удаленных географических мест. С другой стороны, идентичных повторов в одной популяции не было найдено.
Генетические расстояния между 63 нуклеотидными последовательностями pBuM189 были рассчитаны согласно методу двух параметров Кимуры (KIMURA 1980), и был построен дендрограмм «соседнего соединения» (SAITOU и NEI 1987) (Рис. 3). Последовательность Juan/4 находилась на явно отдельной ветви от других повторов pBuM189, что подтверждает предположение о том, что она принадлежит к другой подсемейства pBuM189 satDNA. Повтор Ticu/4 находился на ветви между Juan/4 и оставшимися 61 повтором pBuM189. Среди оставшихся 61 повторов pBuM189 не было наблюдено кластеров, содержащих последовательности pBuM189 из одной популяции или групп географически близких популяций.
В целом, данные указывают на высокие уровни сходства последовательностей pBuM189 среди популяций D. buzzatii, которые расположены, в некоторых случаях, более чем в 3000 км друг от друга. Такое отсутствие межпопуляционной дифференциации satDNA согласуется с данными mtDNA, которые указывают на высокий генетический поток и очень небольшую подструктуру популяций на протяжении большей части ареала D. buzzatii в Южной Америке (ROSSI et al. 1996; DE BRITO et al. 2002). Соответственно, должно быть адаптивное объяснение потери полиморфизма хромосомных инверсий в бразильских популяциях (по сравнению с аргентинскими популяциями), о чем сообщали FIGUEIREDO и SENE (1992).
В настоящее время Чако (северная Аргентина и восточная Боливия) и Каа́тинга (северо-восток Бразилии) являются единственными двумя обширными районами аридной растительности в Южной Америке, представляющими высокую плотность и разнообразие видов кактусов. В других районах кактусы встречаются реже и распределены пятнами. То же самое верно для популяций D. buzzatii, которые зависят от кактусов для питания и размножения. Широко признано, что глобальный климат переживал повторяющиеся циклы холодной-сухой и теплой-влажной погоды в течение плейстоцена, когда произошло как минимум четыре крупных ледниковых события и множество меньших. Например, в последнем ледниковом событии (от 18 000 до 13 000 лет назад) климат в Южной Америке был сухим, и Чако был соединен с Каа́тингой через ксерофитные формации в центральной Бразилии и на Атлантическом побережье (AB'SABER 1977). Следовательно, D. buzzatii вероятно имел гораздо более непрерывное распределение, чем то, что наблюдается сегодня. Такая ситуация могла позволить генетический поток среди южноамериканских популяций D. buzzatii в значительной степени.
На сегодняшний день большинство спутниковых ДНК, описанных в литературе, были изолированы от особей одного штамма или местности. Настоящие данные являются одним из немногих примеров, предоставляющих информацию о вариабельности последовательностей satDNA среди естественных популяций широко распространенного вида.
Авторы: Густаво С. С. Кун, Фернандо Ф. Франко, Уилсон А. Силва мл., Нильсе М. Мартинес-Россі и Фабио М. Сене
Ссылки:
- Аб'Сабер, Н. А. 1977. Пространства, занятые расширением сухих климатов Южной Америки, во время ледниковых периодов четвертичного периода. – Палеоклиматы 3: 1 – 19.
- Бахманн, Л., Рааб, М. и Сперлих, Д. 1989. Спутниковая ДНК и видообразование: специфическая спутниковая ДНК вида Drosophila guanche. – Z. Zool. Syst. Evol. Forsch. 27: 84 – 93.
- Баймай, В., Сене, Ф. М. и Перейра, М. А. Q. Р. 1983. Гетерохроматин и кариотипическая дифференциация некоторых неотропических видов кактусовых муравьев группы Drosophila repleta. – Генетика 67: 81 – 92.
- Баркер, Дж. С. Ф., Сене, Ф. М., Иствуд, П. П. и др. 1985. Аллозимная и хромосомная полиморфизм Drosophila buzzatii в Бразилии и Аргентине. – Генетика 67: 161 – 170.
- Карсон, Х. Л. и Вассерман, М. 1965. Широко распространенный хромосомный полиморфизм у широко распространенного вида Drosophila buzzatii. – Am. Nat. 99: 111 – 115.
- Де Брито, Р. А., Манфрин, М. Х. и Сене, Ф. М. 2002. Филогеография митохондриальной ДНК бразильских популяций Drosophila buzzatii. – Genet. Mol. Biol. 2: 161 – 171.
- Довер, Г. А. 1986. Молекулярный драйв в многогенных семьях: как возникают, распространяются и ассимилируются биологические новшества. – Trends Genet. 2: 159 – 165.
- Элдер, Ф. Дж. и Тёрнер, Б. Дж. 1994. Концертная эволюция на уровне популяции: последовательности спутниковой ДНК HindIII у пупфишей. – Proc. Natl Acad. Sci. 91: 994 – 998.
- Фигейредо, В. Л. С. и Сене, Ф. М. 1992. Вариабельность хромосом в бразильских популяциях Drosophila buzzatii (Diptera, Drosophilidae). – Revta. Bras. Biol. 52: 555 – 561.
- Фонтдевила, А., Руис, А., Окана, Х. и др. 1982. Эволюционная история Drosophila buzzatii. II. Насколько изменился хромосомный полиморфизм при колонизации? – Эволюция 36: 843 – 851.
- Фриденберг, Дж., Пертольди, К., Дальгаард, Дж. и др. 2002. Генетическая вариация в оригинальных и колонизирующих популяциях Drosophila buzzatii, проанализированная с помощью микросателлитных локусов, изолированных с помощью нового метода ПЦР. – Mol. Ecol. 11: 181 – 190.
- Холлибертон, Р. и Баркер, Дж. С. Ф. 1993. Отсутствие вариации митохондриальной ДНК у австралийских Drosophila buzzatii. – Mol. Biol. Evol. 10: 484 – 487.
- Кимура, М. 1980. Единственный метод для оценки эволюционной скорости замены оснований через сравнительные исследования нуклеотидных последовательностей. – J. Mol. Evol. 16: 111 – 120.
- Кинг, Л. М. и Каммингс, М. П. 1997. Вариация последовательности повтора спутниковой ДНК низка у трех видов жуков-кладоносцев рода Nicrophorus (Coleoptera: Silphidae). – Mol. Biol. Evol. 14: 1088 – 1095.
- Кун, Г. С. С., Болльгонн, С., Сперлих, Д. и др. 1999. Характеризация специфической спутниковой ДНК вида Drosophila buzzatii. – J. Zool. Syst. Evol. Res. 37: 109 – 112.
- Кумар, С., Тамура, К., Якобсен, И. Б. и др. 2001. MEGA2: программное обеспечение для молекулярного эволюционного генетического анализа. – Университет штата Аризона.
- Перейра, М. А. Q. Р., Вилела, К. Р. и Сене, Ф. М. 1983. Заметки о местах размножения и кормления некоторых видов группы repleta рода Drosophila (Diptera, Drosophilidae). – Наука и культура 35: 1313 – 1319.
- Россі, М. С., Баррио, Э., Латорре, А. и др. 1996. Эволюционная история Drosophila buzzatii. XXX. Полиморфизм митохондриальной ДНК в оригинальных и колонизирующих популяциях. – Mol. Biol. Evol. 13: 314 – 323.
- Руис, А., Кансиан, А. М., Кун, Г. С. С. и др. 2000. Загадка видообразования Drosophila serido: собираем новые кусочки вместе. – Генетика 108: 217 – 227.
- Сайто, Н. и Ней, М. 1987. Метод соседнего соединения: новый метод для реконструкции филогенетических деревьев. – Mol. Biol. Evol. 4: 406 – 425.
- Тидон-Склорз, Р. и Сене, Ф. М. 1995. Фауна Drosophila (Diptera, Drosophilidae) в северной части «Cadeia do Espinhac¸o», штат Минас-Жерайс и Баия, Бразилия: биогеографические и экологические аспекты. – Iheringia Ser. Zool. 78: 85 – 94.
- Тидон-Склорз, Р., Вилела, К. Р., Сене, Ф. М. и др. 1994. Род Drosophila (Diptera, Drosophilidae) в Серра до Сипо, штат Минас-Жерайс, Бразилия. – Revta. Bras. Ent. 38: 627 – 637.
- Вилела, К. Р., Сене, Ф. М. и Перейра, М. А. Q. Р. 1980. О фауне Drosophila в Чако и восточных склонах Анд в Аргентине. – Revta. Bras. Biol. 40: 837 – 841. Вилела, К. Р., Перейра, М. А. Q. Р. и Сене, Ф. М. 1983. Предварительные данные о географическом распределении видов Drosophila в пределах морфоклиматических доменов Бразилии. II. Группа repleta. – Ciên. Cult. São Paulo 35: 66 – 70.
- Вассерман, М. 1962. Цитологические исследования группы repleta рода Drosophila. V. Подгруппа mulleri. – Univ. Texas Publ. 6205: 85 – 118.
- У, В. Л., Ван, Дж. П., Цзэн, М. Ц. и др. 1999. Клонирование и генетическая вариабельность повторяющейся ДНК HindIII в Acrossocheilus paradoxus (Cyprinidae). – Genome 42: 780 – 788.
