Низкая изменчивость спутниковой ДНК в естественных популяциях Drosophila antonietae, участвующих в различных эволюционных событиях
Машинный перевод
Оригинальная статья написана на языке EN (ссылка для ознакомления) .
Drosophila antonietae является кактусофильным видом, который встречается в мезофильном лесу бассейна реки Парана–Парагвай и на дюнах югоатлантического побережья Бразилии. Хотя генетическая структура популяций бассейна реки Парана–Парагвай уже была установлена, связь между этими популяциями и популяциями на атлантическом побережье остается спорной. В этом исследовании мы сравнили 33 повторяющиеся единицы спутниковой ДНК pBuM-2, изолированные от особей из 8 популяций D. antonietae в этих географических регионах, включая некоторые популяции, найденные в зоне контакта с близкородственной D. serido. Последовательности pBuM-2 показали низкую межпопуляционную изменчивость. Этот результат был интерпретирован как следствие как генетического потока между популяциями, так и неравномерного кроссинговера, способствующего гомогенизации тандемных массивов. Представленные здесь результаты, вместе с результатами предыдущих исследований, подчеркивают использование pBuM-2 для решения таксономических конфликтов внутри кластеров видов D. buzzatii.
Введение
Drosophila antonietae является членом группы видов D. buzzatii, монофилетической группы, состоящей из 7 родственных видов, естественно эндемичных для неотропического региона, встречающихся в открытых и сухих лесах восточной части Южной Америки (Manfrin и Sene 2006). Эти виды используют некротическую ткань кактуса в качестве мест размножения (Pereira et al. 1983); из-за этого их географический ареал ограничен распределением хозяев-какутсов. Таким образом, D. antonietae является привлекательной моделью для изучения эволюционных процессов (Manfrin и Sene 2006).
Географическое распределение D. antonietae можно охарактеризовать двумя способами. В долинах, окруженных бассейном рек Парана–Парагвай, популяции встречаются в мезофильных лесах, ассоциированных с кактусом Cereus hildemannianus (Tidon-Sklorz и Sene 2001; Mateus и Sene 2003). В своем юго-восточном распределении D. antonietae встречается в дюнных районах Атлантического побережья, используя в основном C. hildemannianus, но также кактусы из рода Opuntia (Ruiz et al. 2000; Manfrin и Sene 2006) (Рисунок 1).
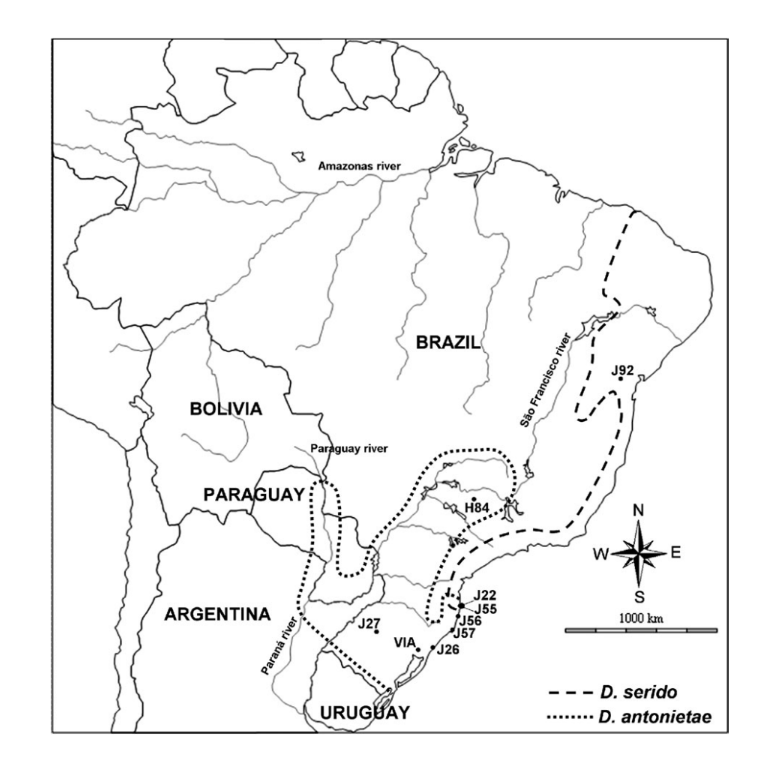
Предыдущие исследования показали, что популяции D. antonietae из бассейна рек Парана–Парагвай структурированы в соответствии с моделью изоляции по расстоянию из-за положительной корреляции морфологических и генетических расстояний с географическими и экологическими расстояниями (последние определяются как расстояние между 2 популяциями вдоль русла реки) (de Brito et al. 2002; Mateus и Sene 2007). Основываясь на числе мигрантов на поколение, Mateus и Sene (2007) предположили умеренное влияние как генетического потока, так и генетического дрейфа в этих популяциях D. antonietae, при этом генетический поток перекрывает генетический дрейф, способствуя образованию генетического градиента.
Филогеографический анализ, основанный на митохондриальной ДНК, показал, что юго-восточное распределение D. antonietae на атлантическом побережье Бразилии было колонизировано в результате событий расширения популяций D. antonietae из центральной депрессии штата Рио Гранде до Сул (de Brito et al. 2002; Manfrin и Sene 2006). Однако этот анализ не смог определить, было ли это расширение сопровождено поддержанием генетического потока с приобретением структуры популяции в соответствии с моделью изоляции по расстоянию или же было последовано фрагментацией, что подразумевает отсутствие генетического потока между популяциями атлантического побережья и популяциями из бассейна рек Парана–Парагвай (de Brito et al. 2002). Данные о хромосомных инверсиях могут указывать на некоторую степень фрагментации между популяциями из этих 2 различных сред, так как популяции из долин бассейна рек Парана–Парагвай полиморфны по инверсиям 2y8, 2z8 и 5e, в то время как популяции с атлантического побережья полиморфны по 2z8 (Ruiz et al. 2000). Популяции D. antonietae на атлантическом побережье имеют еще одну важную характеристику: они находятся в контакте с близкородственным видом D. serido (D. buzzatii кластер) (Ruiz et al. 2000; Manfrin и Sene 2006). Эта зона перекрытия является результатом расширения D. serido с севера на юг атлантического побережья и совместного географического расположения с
D. antonietae популяции (de Brito et al. 2002; Manfrin и Sene 2006) (Рисунок 1). Морфологические признаки и анализ митохондриальной ДНК предполагают, что гибридизация между
D. antonietae и D. serido произошла в зоне контакта (C.K.B. Santos и коллеги, неопубликованные данные).
Сателлитные ДНК — это высокорепетитивные тандемно расположенные последовательности ДНК, которые представляют собой значительную часть гетерохроматина эукариотических организмов (Charlesworth и др. 1tt4; Palomeque и Lorite 2008). Сателлиты обычно расположены рядом с центромерными регионами и, реже, в теломерах (Ugarkovic и Plohl 2002; Kuhn и др. 2008). Эти последовательности эволюционируют согласованно, в то время как основные молекулярные механизмы, участвующие в их эволюции, — это репликация сдвига, неравномерный кроссинговер, конверсия генов и репликация по кругу (Dover 1982; Charlesworth и др. 1994).
Некоторые из семейств спутниковой ДНК быстро эволюционируют, что позволяет использовать их в качестве молекулярных маркеров в межвидовых сравнительных исследованиях (Watabe et al. 1997; Picariello et al. 2002; Kuhn and Sene 2005). Однако использование этих последовательностей в качестве молекулярных маркеров в популяционных исследованиях было относительно недостаточно изучено, несмотря на то, что консервативная эволюция была зарегистрирована в популяциях Cyprinodon variegates (Cyprinodontidae) (Elder and Turner 1994) и Acrossocheilus paradoxus (Cyprinidae) (Wu et al. 1999).
Три семейства спутниковой ДНК, общие для видов кластера D. buzzatii, были формально описаны: pBuM (Kuhn et al. 1999, 2008; Kuhn and Sene 2005), DBC-150 (Kuhn et al. 2007), и SSS139 (Franco et al. 2008). Семейство pBuM состоит из 2 подсемейств, pBuM-1 и pBuM-2 (Kuhn and Sene 2005; Kuhn et al. 2008). Подсемейство pBuM-2 состоит из AT-богатых мономеров длиной 370 п.н., которые имеют качественные (фиксированная замена нуклеотидов) и количественные (число копий) различия среди видов кластера D. buzzatii (Kuhn and Sene 2005; Kuhn et al. 2007, 2008).
В этой работе мы демонстрируем интраспецифическую вариабельность спутниковой ДНК pBuM-2 в D. antonietae, используя популяции из 2 различных сред (бассейн Парана–Парагвай и Атлантическое побережье), которые имеют различные эволюционные истории, связанные с поддержанием генетического потока в первой и событиями гибридизации с близкородственным видом D. serido во второй. Мы выбрали спутниковую ДНК pBuM в качестве молекулярного маркера, потому что она демонстрирует 14 диагностических нуклеотидных замен между D. antonietae и D. serido (Kuhn и Sene 2005). Таким образом, pBuM позволяет таксономическую идентификацию образцов, изолированных из зоны контакта. Более того, поскольку эволюционная динамика последовательностей спутниковой ДНК на интраспецифическом уровне не полностью понята, дальнейшие эмпирические данные могут способствовать лучшему пониманию процесса, лежащего в основе эволюции спутниковой ДНК (Ugarkovic и Plohl 2002; Palomeque и Lorite 2008).
Материалы и методы
Образцы
Мы изучили 8 бразильских популяций D. antonietae (Рисунок 1). Три из этих популяций расположены в долинах бассейна реки Парана–Парагвай: J2/ (Сантьяго, штат Риу-Гранди-ду-Сул), Via (Виамão, штат Риу-Гранди-ду-Сул) и H84 (Серрана, штат Сан-Паулу, номер доступа AY656609– AY656615; Kuhn и Sene 2005); и 5 расположены на дюнах Атлантического побережья, в зоне контакта с
D. serido: J56 (Гаропаба, штат Санта-Катарина), J55 (пляж Армакан, Флорианополис, штат Санта-Катарина), J22 (пляж Жоакина, Флорианополис, штат Санта-Катарина), J5/ (Лагуна, штат Санта-Катарина) и J26 (Осорио, штат Риу-Гранди-ду-Сул) (Рисунок 1). Последовательности D. antonietae анализировались вместе с последовательностями G pBuM-2 из D. serido (популяция Jf2—Милагрес, штат Баия, номер доступа AY656616–AY656621; Kuhn и Sene 2005), чтобы выявить некоторые события интрогрессии в популяциях с Атлантического побережья.
Изоляция спутниковой ДНК pBuM-2 и анализ последовательности
Геномная ДНК от одного самца (пойманные в дикой природе мухи) из каждой популяции была извлечена с использованием набора для очистки геномной ДНК Wizard (Promega). Геномная ДНК была подвергнута полимеразной цепной реакции для амплификации копий семейства pBuM-2 [праймеры A2F (CGGAGTA-TTTTTCATTCGAC) и A2R (GGTATGCCATAAAG-AAGTCG)] в соответствии с Kuhn et al. (2008). Полученные полосы длиной примерно 400 п.н. были элюированы из геля путем инкубации на ночь в элюционной жидкости (500 мМ NaAc; 1 мМ этилендиаминтетрауксусной кислоты). Восстановленные фрагменты были клонированы с использованием набора для клонирования с тупыми концами pMOSBlue (RPN 5110; Amersham Pharmacia Biotech). Плазмидная ДНК была подготовлена в соответствии с методологией, описанной в Sambrook et al. (1989), и реакция с ДНК-шаблоном для секвенирования была подготовлена в соответствии с руководством по набору готовых реакций BigDye Terminator Cycle Sequencing (PerkinElmer). Автоматическое секвенирование ДНК было выполнено на секвенаторе ABI Prism 377 (PerkinElmer).
Сравнения последовательностей были выполнены в CLUSTALW 1.8 (Thompson et al. 1994) и отредактированы в BioEdit (Hall 1999). Мы использовали алгоритм и статистическую модель, предложенные Betrán et al. (1997), для обнаружения участков генной конверсии. Параметр Rm, который оценивает минимальное количество событий рекомбинации в образце ДНК (Hudson and Kaplan 1985), использовался для выявления событий рекомбинации. Хотя этот параметр не измеряет неравномерный кроссинговер, разумно предположить, что Rm будет иметь значения выше нуля, если неравномерный кроссинговер имел место. В этом смысле мы используем Rm как косвенное доказательство неравномерного кроссинговера. Генетические расстояния были рассчитаны согласно p-расстоянию, которое является пропорцией нуклеотидных различий между 2 последовательностями (т.е. p 5 Np/N, где Np — различия нуклеотидов, а N — общее количество нуклеотидов). Более сложные модели расстояний также применялись к данным, с аналогичными результатами, как и те, что представлены по p-расстоянию (данные не показаны). Матрица расстояний использовалась для создания фенограммы с использованием метода соседнего соединения (Saitou and Nei 1987). Расчеты генетических расстояний и фенограммы были выполнены с использованием программы MEGA 3.0 (Kumar et al. 2004).
Мы также исследовали паттерны внутривидовой вариации в D. antonietae с помощью анализа молекулярной вариации (AMOVAs; Excoffier et al. 1992). Для AMOVA популяции D. antonietae были сгруппированы на 2 группы: Атлантическое побережье и бассейн рек Парана–Парагвай. AMOVA была выполнена с использованием Arlequin 3.0 (Excoffier et al. 2005).
Результаты и обсуждение
Было получено двадцать шесть новых мономеров спутниковой ДНК pBuM-2: 4 из местности J56 (номер доступа FJ935973– FJ93597G); 4 из местности Via (номер доступа FJt35t77–FJt35t80); 4 из местности J26 (номер доступа FJ935981–FJ935984); 3 из местности J5/ (номер доступа FJ935985–FJ935987); 5 из местности J2/ (номер доступа FJ935988–FJ935992); 2 из местности J55 (номер доступа FJ935993–FJ935994); и 4 из местности J22 (номер доступа FJ935995–FJ935998). Высокая нуклеотидная схожесть между последовательностями, полученными в этой работе, и ранее описанными (Kuhn and Sene 2005) подтверждает, что они принадлежат к семейству спутниковой ДНК pBuM-2.
Все полученные последовательности представляют собой первичную структуру, типичную для D. antonietae, и были сгруппированы в одну ветвь в фенограмме, вместе с ранее описанными последовательностями pBuM-2 D. antonietae (Рисунок 2), даже те, что из популяций в зоне контакта с D. serido, где предполагалась гибридизация между D. antonietae и D. serido с интрогрессией митохондриальной ДНК (C.K.B Santos, F.M. Sene и M.H. Manfrin, неопубликованные данные). Эта ситуация может быть связана с наблюдениями, что митохондриальные гены, как правило, интрогрессируют легче, чем ядерные последовательности (Dorado et al. 1992; Arnold 1993). Наиболее часто упоминаемые объяснения для этого наблюдения связаны с процессами цитоядерного дисбаланса (Arnold 1993) и стерильностью гибридных самцов (Aubert и Solignac 1990).
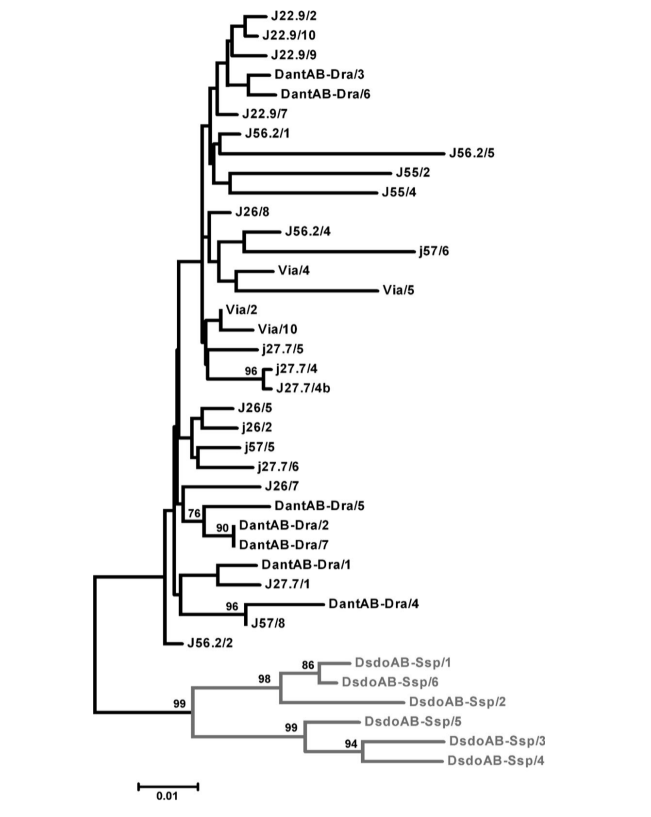
Существует высокий уровень последовательного сходства среди последовательностей pBuM-2 D. antonietae (в среднем 95.7%), и не было найдено мутаций, которые могли бы служить диагностическими для конкретной популяции. Межпопуляционная изменчивость была очень похожа на внутрипопуляционную изменчивость (Таблица 1), и последовательности из одной и той же популяции не группировались вместе в конкретной ветви дендрограммы (Рисунок 2). AMOVA, проведенный с популяциями, разделенными по географическим группам (бассейн реки Парана–Парагвай и Атлантическое побережье), показал, что 99.30% общей генетической изменчивости можно отнести к внутригрупповой изменчивости, в то время как всего 0.70% было отнесено к межгрупповой изменчивости (Таблица 2). Рассчитанное значение Фст было низким и не имело статистической поддержки (Таблица 2).
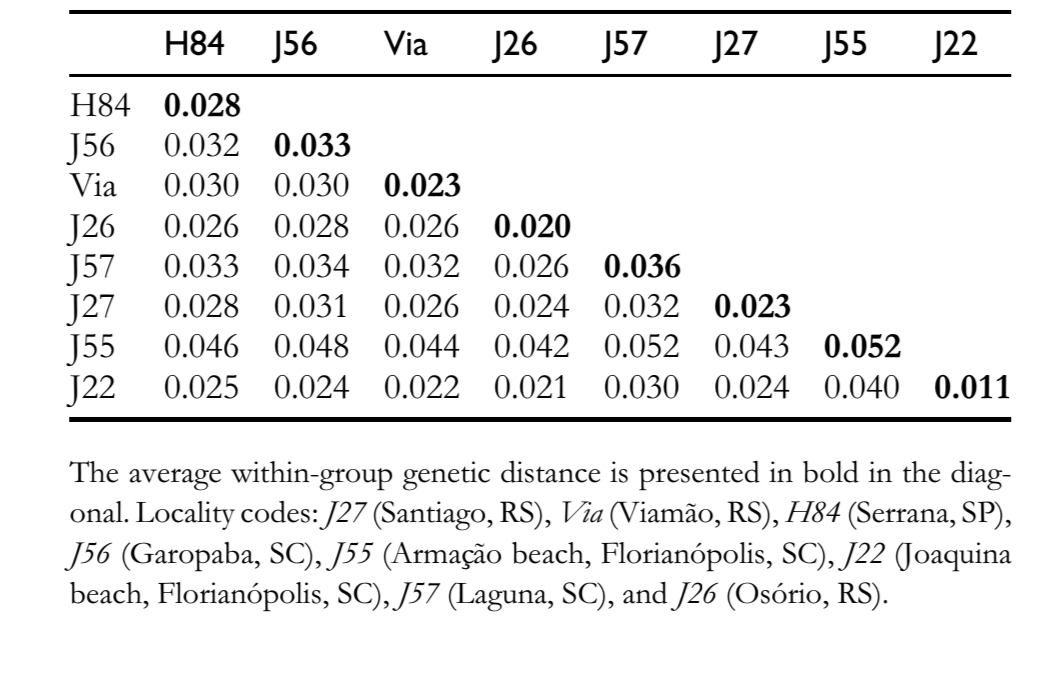

Популяции D. antonietae из бассейна рек Парана–Парагвай структурированы в соответствии с моделью изоляции по расстоянию (Монтейро и Сене 1995; Матеус и Сене 2007). Наблюдаемые хромосомные инверсии указывают на фрагментацию между популяциями с Атлантического побережья и популяциями из бассейна рек Парана–Парагвай (Руис и др. 2000), в то время как данные митохондриальной ДНК не смогли определить, была ли фрагментация или изоляция по расстоянию доминирующей структурой, определяющей взаимосвязь этих 2 групп популяций (де Брито и др. 2002). Высокая нуклеотидная схожесть pBuM-2 в D. antonietae (Рисунок 2 и Таблицы 1 и 2) согласуется с поддержанием генного потока между популяциями этого вида, даже среди тех, которые находятся в бассейне рек Парана–Парагвай и на Атлантическом побережье. Наши результаты предполагают, что изоляция по расстоянию должна объяснять вариабельность митохондриальной ДНК в D. antonietae; эта модель предсказывает генетический поток между популяциями, как это было выведено на основе данных о спутниковой ДНК, представленных здесь. В этом контексте наименьшие полиморфизмы хромосомных инверсий в популяциях с Атлантического побережья D. antonietae, обнаруженные Руисом и др. (2000), могут быть объяснены как следствие исторических и демографических событий, таких как расширение популяции из популяций долин бассейна рек Парана–Парагвай, как это предполагается на основе митохондриальной ДНК (де Брито и др. 2002). Альтернативное объяснение этой дифференциации популяций может быть связано с местной адаптацией к различным условиям окружающей среды при наличии генного потока, так как хромосомные инверсии могут быть связаны с адаптивными характеристиками у кактусофильных Drosophila (Хассон и др. 1992; Фернандес Ириарт и др. 2003). Альтернативное объяснение высокой нуклеотидной консервации pBuM-2 в D. antonietae основано на эволюционной динамике последовательностей спутниковой ДНК. Молекулярные механизмы, связанные с гомогенизацией последовательно расположенных последовательностей, такие как неравномерный кроссинговер и генетическая конверсия через процесс, называемый молекулярным драйвом (Довер 1982; Стрэхан и др. 1985), могут действовать на спутниковую ДНК pBuM у этого вида. Наши анализы не выявили участков генетической конверсии в последовательностях pBuM-2, изолированных от D. antonietae, что предполагает, что этот молекулярный механизм не влияет на разнообразие последовательностей pBuM-2 у этого вида. Однако было обнаружено минимум 4 события рекомбинации в образце (Rm = 4) между интервалами 130–136, 136–155, 174–376 и 376–378. Эти события рекомбинации могут быть косвенным доказательством неравномерного кроссинговера, способствующего поддержанию первичной структуры последовательностей pBuM-2 в различных популяциях D. antonietae. В соответствии с этой гипотезой, геном D. antonietae содержит большое количество мономеров pBuM-2 (Кун и др. 2007; Кун и др. 2008), что увеличивает вероятность неравномерного кроссинговера (Смит1976), и, следовательно, гомогенизации и согласованной эволюции.
Эти 2 альтернативные гипотезы, объясняющие высокую нуклеотидную схожесть, обнаруженную в мономерах pBuM-2 D. antonietae, не являются взаимоисключающими. В этом контексте мы предполагаем, что баланс между генетическим потоком среди популяций и событиями гомогенизации тандемных массивов спутниковой ДНК, способствованными неравным кроссинговером, способствует сохранению первичной структуры pBuM-2 в видах D. antonietae.
Отсутствие молекулярной дивергенции в последовательностях спутниковой ДНК между популяциями одного и того же вида, как показано в настоящей статье, также было отмечено у других видов насекомых (Bachmann et al. 1994, 1998; Lorite et al. 2002; Feliciello et al. 2005). Например, Bachmann et al. (1994) обнаружили высокие уровни генетической схожести среди мономеров спутниковой ДНК pDoP102 в популяциях Dolichopoda schiavazzii (Orthoptera), которые, согласно аллозиматическим данным, были изолированы друг от друга как минимум на протяжении 235 000 лет. По мнению этих авторов, однородность в pDoP102 даже в изолированных популяциях может быть связана с биологией спутниковой ДНК, поскольку неравный кроссинговер и генетическая конверсия могут способствовать поддержанию первичной структуры последовательностей спутниковой ДНК (Bachmann et al. 1994). В другом исследовании наличие участков генетической конверсии в мономерах семейства спутниковой ДНК использовалось как доказательство событий гомогенизации в этой спутниковой ДНК, объясняя высокую интраспецифическую сохранность спутниковой ДНК, обнаруженную у фитофагового жука Xanthogaleruca luteola (Lorite et al. 2002).
В кластерном виде D. buzzatii также была выявлена высокая интраспецифическая сохранность последовательности pBuM-2 в D. seriema (Kuhn и Sene 2004) и D. gouveai (Franco, Kuhn и др. 2006). Более того, популяции D. buzzatii являются однородными по отношению к спутниковой ДНК pBuM-1, еще одному члену семейства pBuM (Kuhn и др. 2003). В D. seriema и D. buzzatii отсутствие дифференциации популяций в последовательностях спутниковой ДНК было предложено как результат прошлых и текущих событий генетического потока между популяциями, что согласуется с результатами, полученными от других генетических маркеров, анализируемых в тех же популяциях (Kuhn и др. 2003; Kuhn и Sene 2004). Для
D. gouveai, который структурирован согласно модели «остров» (de Brito et al. 2002; Moraes и Sene 2007), основная гипотеза, объясняющая сохранение первичной структуры в спутниковой ДНК pBuM-2, заключалась в том, что независимые механизмы гомогенизации, способствующие повторяющимся единицам, общим для популяций, произошли после события изоляции популяций D. gouveai (Franco, Kuhn и др. 2006). Кластер D. buzzatii состоит из родственных видов, таксономия которых основана на морфометрических характеристиках адеагуса, который является интромитентным органом мужских половых органов насекомых (Vilela 1983; Tidon-Sklorz и Sene 2001; Franco, Prado и др. 2006). Таким образом, идентификация самок кластера D. buzzatii, полученных из естественной среды, осуществляется путем идентификации их мужских потомков, что делает необходимым создание изофемальных линий, что может быть трудной задачей для кактусофильных Drosophila видов. Более того, правильная дискриминация видов, основанная только на количественных признаках, таких как морфология адеагуса, иногда затруднена из-за наличия морфологически неоднозначных особей, особенно когда особи собираются в зонах контакта между видами кластера D. buzzatii. В этом смысле сильное внутривидовое сохранение последовательностей спутниковой ДНК pBuM, полученных в нескольких исследованиях (Kuhn et al. 2003; Kuhn и Sene 2004; Franco, Kuhn и др. 2006; настоящая работа), даже в генетически структурированных видах, подчеркивает полезность pBuM-2 в решении таксономических конфликтов внутри видов кластера D. buzzatii, признанной модели для исследований в области эволюционной биологии (Manfrin и Sene 2006).
Авторы: Фернандо Фария Франко, Фабио Мело Сене, Маура Хелена Манфрин
Ссылки:
- Арнольд Дж. 1993. Цитонуклеарные дисбалансы в гибридных зонах. Annu Rev Ecol Syst. 24:521–554.
- Оберт Ж, Солиньяк М. 1990. Экспериментальные доказательства интрогрессии митохондриальной ДНК между видами Drosophila. Эволюция. 44:1272–1282.
- Бахманн Л, Томиук Ж, Адис Ж, Вохланд К. 1998. Генетическая дифференциация многоножки Pycnotropis epiclysmus, обитающей в сезонно затопляемых и незатопленных амазонских лесах. J Zool Syst Evol Res. 36:65–70.
- Бахманн Л, Венанцетти Ф, Сбордони В. 1994. Характеризация семейства специфической спутниковой ДНК Dolichopoda schiavazzi (Orthoptera, Rhaphidophoridae) пещерных сверчков. J Mol Evol. 39:274–281.
- Бетран Э, Розас Ж, Наварро А, Барбадилья А. 1997. Оценка числа и распределения длины участков генной конверсии на основе данных последовательностей ДНК популяции. Генетика. 146:89–99.
- Чарльзворт Б, Снеговски П, Штефан В. 1tt4. Эволюционная динамика повторяющейся ДНК у эукариот. Природа. 371:215–220.
- де Брито АР, Манфрин МХ, Сене ФМ. 2002. Нестедная кладистическая анализ бразильских популяций Drosophila serido. Mol Phylogenet Evol. 22: 131–143.
- Дорадо О, Ризеберг ЛХ, Ариас Д. 1992. Интрогрессия хлоропластной ДНК в подсолнечниках южной Калифорнии. Эволюция. 46:566–572.
- Довер Г. 1982. Молекулярный драйв: когезионный режим эволюции видов. Природа. 299:111–117.
- Элдер ДжФ, Тёрнер БД. 1tt4. Концертированная эволюция на уровне популяции: спутниковые ДНК последовательности HindIII у рыб-пупков. Proc Natl Acad Sci U S A. 91: 994–998.
- Эккоффье Л, Лаваль Г, Шнайдер С. 2005. Arlequin (версия 3.0): интегрированный программный пакет для анализа данных популяционной генетики. Evol Bioinform Online. 1:47–50.
- Эккоффье Л, Смус ПЕ, Кваттро ЖМ. 1992. Анализ молекулярной вариации, выведенной из метрических расстояний между гаплотипами ДНК: применение к данным ограничения митохондриальной ДНК человека. Генетика. 131:479–491.
- Феличиелло И, Пикариелло О, Чинали Г. 2005. Первая характеристика общей изменчивости повторяющихся единиц в виде показывает неожиданные особенности спутниковой ДНК. Ген. 349:153–164.
- Фернандес Ириарте ПЖ, Норри ФМ, Хассон ЕР. 2003. Хромосомные инверсии влияют на размер и форму тела в различных размножительных ресурсах у Drosophila buzzatii. Наследственность. 91:51–59.
- Франко ФФ, Кун ГЦС, Сене ФМ, Манфрин МХ. 2006. Сохранение последовательностей спутниковой ДНК pBuM–2 среди географически изолированных популяций Drosophila gouveai из Бразилии. Генетика. 128:287–295.
- Франко ФФ, Прадо ПРР, Сене ФМ, Коста ЛФ, Манфрин МХ. 200G. Морфология адеагуса как дискриминантный маркер у двух близкородственных кактусофильных видов Drosophila (Diptera; Drosophilidae) в Южной Америке. An Acad Bras Cienc. 78:203–212.
- Франко ФФ, Сене ФМ, Манфрин МХ. 2008. Молекулярная характеристика семейства спутниковой ДНК SSS13t у сестринских видов кластера Drosophila buzzatii. Genet Mol Biol. 31:155–159.
- Холл ТА. 1999. BioEdit: удобный редактор выравнивания биологических последовательностей и программа анализа для Windows t5/t8/NT. Nucleic Acids Symp Ser. 41:95–98.
- Хассон Е, Фанара ДжД, Родригес С, Виларди ДжК, Рейг ОА, Фонтдевила А. 1992. Эволюционная история Drosophila buzzatii. XXIV. Инверсии второй хромосомы имеют различный средний эффект на длину груди. Наследственность. 68:557–563.
- Хадсон РР, Каплан НЛ. 1985. Статистические свойства числа рекомбинационных событий в истории выборки последовательностей ДНК. Генетика. 111:147–164.
- Кун ГЦС, Болльгонн С, Сперлич Д, Бахманн Л. 1999. Характеризация специфической спутниковой ДНК Drosophila buzzatii. J Zool Syst Evol Res. 37:109–112.
- Кун ГЦС, Франко ФФ, Манфрин МХ, Морея-Фильо О, Сене ФМ. 2007. Низкие темпы гомогенизации семейства спутниковой ДНК DBC-150, ограниченного одной парой микрохромосом у видов из кластера Drosophila buzzatii. Chromosome Res. 15:457–469.
- Кун ГЦС, Франко ФФ, Силва ВА мл, Мартинес-Россі НМ, Сене ФМ. 2003. О вариабельности спутниковой ДНК pBuM18t среди южноамериканских популяций Drosophila buzzatii. Наследственность. 139:161–166.
- Кун ГЦС, Сене ФМ. 2004. Характеризация и межпопуляционная изменчивость сложной спутниковой ДНК HpaI у Drosophila seriema (группа repleta). Генетика. 121:241–249.
- Кун ГЦС, Сене ФМ. 2005. Эволюционный оборот двух подсемейств спутниковой ДНК pBuM в кластере видов Drosophila buzzatii (группа repleta): от alpha до alpha/beta массивов. Ген. 349:77–85.
- Кун ГЦС, Сене ФМ, Морея-Фильо О, Шварцачер Т, Хеслоп-Харрисон ДжС. 2008. Анализ последовательностей, хромосомное распределение и организация на больших расстояниях показывают, что быстрый оборот новых и старых повторов спутниковой ДНК pBuM приводит к различным паттернам вариации у семи видов кластера Drosophila buzzatii. Chromosome Res. 16:307–324.
- Кумар С, Тамура К, Ней М. 2004. MEGA3: интегрированное программное обеспечение для анализа молекулярной эволюционной генетики и выравнивания последовательностей. Brief Bioinform. 5:150–163.
- Лорите П, Каррильо ХА, Гарнерия И, Пети́пьер Э, Паломе́ке Т. 2002. Спутниковая ДНК у жука-листовика, Xanthogaleruca luteola (Coleoptera, Chrysomelidae): характеристика, межпопуляционный анализ и расположение хромосом. Cytogenet Genome Res. 98:302–307.
- Манфрин МХ, Сене ФМ. 200G. Кактусофильные Drosophila в Южной Америке: модель для эволюционных исследований. Генетика. 126:57–75.
- Матейуш РП, Сене ФМ. 2003. Временная и пространственная аллозимная изменчивость у южноамериканской кактусофильной Drosophila antonietae (Diptera; Drosophilidae). Biochem Genet. 41:219–233.
- Матейуш РП, Сене ФМ. 2007. Популяционно-генетическое исследование аллозимной изменчивости в естественных популяциях Drosophila antonietae (Insecta, Diptera). J Zool Syst Evol Res. 45:136–143.
- Монтейро СГ, Сене ФМ. 1tt5. Морфометрическое исследование популяций Drosophila serido из Центрального и Южного Бразилии. Rev Bras Genet (Suppl). 18:283.
- Мораес ЭМ, Сене ФМ. 2007. Микросателлитная и морфометрическая изменчивость у Drosophila gouveai: относительная важность исторических и современных факторов в формировании генетической структуры популяции. J Zoolog Syst Evol Res. 45:336–344.
- Паломе́ке Т, Лорите П. 2008. Спутниковая ДНК у насекомых: обзор. Наследственность. 100:564–573.
- Перейра МАQR, Вилела КР, Сене ФМ. 1983. Заметки о местах размножения и кормления некоторых видов группы repleta рода Drosophila (Diptera, Drosophilidae). Cienc Cult. 35:1313–1319.
- Пикариелло О, Феличиелло И, Беллинеро Р, Чинали Г. 2002. Спутниковая ДНК S1 как таксономический маркер у коричневых лягушек: молекулярные доказательства того, что Rana graeca graeca и Rana graeca italica являются разными видами. Геном. 45: 63–70.
- Руис А, Кассиан АМ, Кун ГЦС, Алвес МАР, Сене ФМ. 2000. Загадка специирования Drosophila serido: собираем новые кусочки вместе. Генетика. 108:217–227.
- Сайто Н, Ней М. 1987. Метод соседнего соединения: новый метод для реконструкции филогенетических деревьев. Mol Biol Evol. 4:406–425.
- Сэмбрук Дж, Фритш ЭФ, Маниатис Т. 1989. Молекулярное клонирование: лабораторный справочник. 2-е изд. Холодный источник Харбор (Нью-Йорк): Издательство лаборатории Холодного источника.
- Смит ГП. 1976. Эволюция повторяющихся последовательностей ДНК путем неравного кроссовера. Наука. 191:528–535.
- Страхан Т, Уэбб Д, Довер ГА. 1985. Переходные стадии молекулярного драйва в многокопийных ДНК-семьях у Drosophila. EMBO J. 4:1701–1708.
- Томпсон ДжД, Хиггинс ДГ, Гибсон ТД. 1994. CLUSTAL W: улучшение чувствительности прогрессивного множественного выравнивания последовательностей за счет взвешивания последовательностей, штрафов за пробелы, специфичных для позиций, и выбора весовой матрицы. Nucleic Acids Res. 22:4673–4680.
- Тидон-Склорц Р, Сене ФМ. 2001. Два новых вида из сестринского набора Drosophila serido (Diptera, Drosophilidae). Iheringia. 90:141–146.
- Угаркович Д, Плохл М. 2002. Изменчивость профилей спутниковой ДНК — причины и последствия. EMBO J. 21:5955–5959.
- Вилела КР. 1983. Ревизия группы видов Drosophila repleta (Diptera; Drosophilidae). Rev Bras Entomol. 27:1–114.
- Ватабе Х, Бахманн Л, Харинг Е, Сперлич Д. 1997. Таксономические и молекулярные исследования Drosophila sinobscura и D. hubeiensis, двух сестринских видов группы D. obscura. J Zool Syst Evol Res. 35:81–94.
- У WL, Ванг JP, Цзэн MC, Цзян TY. 1999. Клонирование и генетическая изменчивость повторяющейся ДНК HindIII в Acrossocheilus paradoxos (Cyprinidae). Геном. 42:780–788.
